Artigo
|
|
| Análise química de méis de abelhas sem ferrão nativas do brasil e suas bebidas fermentadas Chemical analysis of honeys from stingless bees native to brazil and their fermented beverages |
|
Juliana M. FeresI; Luís Guilherme P. FeitosaII; Larissa G. EliasI; Tulio Marcos NunesI; Norberto P. LopesII,* I Casa Melí Ltda., 13560-647 São Carlos - SP, Brasil Recebido: 06/06/2025; *e-mail: npelopes@fcfrp.usp.br Editor responsável pelo artigo: Jorge M. David This study investigated the chemical composition of honeys produced by three species of native stingless bees of Brazil (Tetragonisca angustula, Melipona quadrifasciata, and Scaptotrigona depilis) and their respective fermented beverages (meads) using metabolomic approaches. The honeys were subjected to spontaneous fermentation, as well as fermentation with commercial yeasts. Spontaneous fermentation occurred from microorganisms already present in the honey. In commercial fermentation, the honeys were sterilized and inoculated with selected yeast. Chemical characterization was performed by high-performance liquid chromatography coupled to tandem mass spectrometry with electrospray ionization (HPLC-ESI-MS/MS) and molecular network analysis using the Global Natural Products Social Molecular Networking (GNPS) platform, and by gas chromatography (GC-MS) to evaluate volatile compounds. It was observed that the annotated flavonoids, phenolamides, and glycosylated amino acids were preserved even after the fermentation process. Furthermore, the fermentation products presented a wide diversity of volatile compounds, with variations between species and fermentation processes. The results highlight the potential of honeys and meads as sources of bioactive compounds, contributing to the valorization of Brazilian biodiversity and possibilities for innovation in the fermented beverage market, as well as applications in functional foods and pharmaceutical products. INTRODUÇÃO As abelhas sem ferrão (Apidae, tribo Meliponini), também chamadas de nativas, abelhas indígenas sem ferrão, ou meliponíneos,1,2 representam o maior grupo de abelhas eussociais da Terra, apresentando distribuição pantropical.3 Entre as mais de 400 espécies de abelhas sem ferrão (ASF) descritas na região Neotropical, mais de 240 são registradas no Brasil, as quais pertencem a 29 gêneros, sendo que mais de 80 destas espécies são endêmicas do território brasileiro.4 Essas abelhas diferem em seus comportamentos, comunicação química, forrageamento, estratégias de defesa, arquitetura de ninhos, tamanhos,1,2 sendo um grupo bastante diversificado. Para o estudo selecionamos três espécies de abelhas nativas sem ferrão, Tetragonisca angustula popularmente conhecida como Jataí, a Melipona quadrifasciata, conhecida como Mandaçaia, e a Scaptotrigona depilis, a Mandaguari. A escolha das espécies foi definida de acordo com sua distribuição geográfica, sua popularidade entre os criadores de abelhas nativas e a presença de seus produtos no mercado. O mel é um dos produtos naturais mais apreciados e valorizados pela humanidade devido às suas propriedades nutricionais e medicinais. O mel contém uma variedade de compostos bioativos que contribuem para suas propriedades terapêuticas.5 Entre os principais grupos de compostos encontrados no mel estão os flavonoides, fenólicos, ácidos orgânicos e enzimas como a diastase e a glicose oxidase.5 Estes compostos, principalmente os flavonoides, são responsáveis por atividades antioxidantes, se ligando a radicais livres e neutralizando-os, além de apresentar ação antimicrobiana e anti-inflamatória.6-8 O mel é uma substância produzida por abelhas a partir do néctar das plantas, sendo o mel de abelhas melíferas (Apis mellifera) o mais conhecido e consumido mundialmente. Entretanto, diversas outras espécies de abelhas, conhecidas como abelhas sem ferrão (ASF) (tribo Meliponini) também produzem méis que vêm ganhando popularidade principalmente por apresentarem perfis sensoriais singulares.1,9 Os méis de ASF apresentam características físico-químicas distintas daquele das abelhas A. mellifera, como maior teor de umidade e uma microbiota própria, tornando-o naturalmente mais suscetível à fermentação, o que enriquece sua diversidade de compostos químicos e aumenta sua complexidade sensorial.10,11 O aumento da disponibilidade e do consumo do mel das ASF tem levantado o interesse a respeito desses produtos oriundos da biodiversidade brasileira. A composição química dos méis de abelhas sem ferrão é bastante complexa, podendo ser influenciada pelas suas origens botânicas e geográficas, bem como pelas condições ambientais e espécie de abelha.12 O mel é produzido a partir do néctar floral coletado por essas abelhas e modificado por secreções salivares, das glândulas do abdômen e enzimas de glândulas cefálicas.12 Na composição micromolecular dos méis produzidos por ASF, nota-se o predomínio de fenólicos, como flavonoides, ácidos fenólicos e derivados hidroxicinâmicos.13,14 Além do consumo de mel in natura, uma alternativa para a valorização desse produto é a produção de bebidas alcoólicas fermentadas à base de mel, conhecidas como hidroméis. O hidromel é amplamente reconhecido como uma das bebidas alcoólicas mais antigas do mundo, com evidências de sua produção datando de aproximadamente 7.000 a.C. na China.15 Diversas culturas ao longo da história, como os gregos, romanos, celtas, vikings e egípcios, adotaram a produção e o consumo de hidromel.16 Tradicionalmente, o hidromel é obtido pela fermentação de uma mistura de mel, água e leveduras. Sua receita pode ser enriquecida com uma variedade de ingredientes adicionais, como frutas (maçãs, amoras, cerejas), especiarias (canela, cravo, gengibre) e ervas (tomilho, alecrim), resultando em uma ampla gama de variações de sabor e estilo.16 Nos últimos anos, o hidromel tem experimentado um ressurgimento significativo de popularidade, especialmente nos Estados Unidos e na Europa.17 Nos EUA, o número de hidromelarias cresceu impressionantes 650%, passando de 60 em 2003 para 450 em 2020, refletindo um crescente interesse dos consumidores por bebidas artesanais e tradicionais.18 Na Europa, o mercado de hidromel também tem se expandido, com um aumento na realização de festivais e competições dedicadas à bebida. O renascimento global dessa bebida oferece uma oportunidade única para o desenvolvimento de novos produtos a partir do mel das ASF encontradas no Brasil. O mel produzido por essas abelhas é conhecido por sua diversidade e características sensoriais complexas, proporcionando um perfil único para a produção de hidroméis. Além de contribuir para a inovação no mercado de bebidas, a popularização dos méis das ASF fortalece a valorização da biodiversidade do Brasil, promovendo a conservação e o uso sustentável da biodiversidade brasileira. Este estudo teve como objetivo caracterizar a composição química dos méis de três espécies de ASF encontradas no Brasil e de seus respectivos hidroméis, utilizando uma abordagem metabolômica. Para isso, foram empregadas técnicas analíticas hifenadas - cromatografia gasosa acoplada à espectrometria de massas (CG-EM) e cromatografia líquida de alta eficiência acoplada à espectrometria de massas em tandem com ionização por electrospray (CLAE‑IES‑EM/EM) - permitindo uma análise abrangente de metabólitos de diferentes polaridades. Os dados obtidos por CLAE-IES-EM/EM foram explorados por meio da plataforma GNPS (Global Natural Products Social Molecular Networking), que possibilita a construção de redes moleculares baseadas na similaridade de fragmentação espectral, mesmo na ausência de padrões analíticos.19-22 Essa ferramenta facilita a organização, anotação e interpretação de grandes conjuntos de dados, permitindo a identificação de núcleos químicos compartilhados e compostos putativamente bioativos, chegado até mesmo a identificação de microrganismos nessas matrizes.23 Estudos anteriores,24 visando aprimorar a análise de alimentos por ferramentas de redes moleculares, indicaram que os méis comerciais das espécies desse estudo apresentavam uma assinatura química que poderia permitir seu reconhecimento na dieta humana. Em resumo, os resultados que buscávamos com o presente artigo, utilizando as estratégias citadas, ofereceram uma visão integrada da diversidade química dos méis e hidroméis analisados, com implicações para a identificação de marcadores químicos, avaliação sensorial e segurança alimentar. A caracterização obtida também fornece subsídios científicos para a valorização de produtos derivados da biodiversidade brasileira, fortalecendo o reconhecimento comercial das abelhas nativas e promovendo a conservação dos biomas onde elas atuam.
PARTE EXPERIMENTAL Obtenção dos méis in natura Méis produzidos pelas espécies Tetragonisca angustula, Melipona quadrifasciata e Scaptotrigona depilis foram coletados em outubro de 2021 no município de São Carlos, estado de São Paulo, Brasil (-22,08509, -47,82868). Os méis foram coletados in natura diretamente a partir dos potes de mel de ninhos de um meliponário de criação racional. A coleta de amostras dos méis foi realizada com seringa, sendo que estas amostras foram armazenadas em microtubos sob refrigeração até as análises. Obtenção dos produtos fermentados O processo de obtenção do mosto foi idêntico para os três tipos de méis utilizados nas fermentações. Inicialmente, os méis in natura foram individualmente homogeneizados em recipientes estéreis. As receitas usaram mel na proporção 1:3 (m v-1), com 250 g de mel para 1 L de água. Após misturar e medir os açúcares dissolvidos com um refratômetro, o mel foi adicionado ao mosto até alcançar 23 °Bx, resultando em uma bebida com cerca de 11% de álcool. Após a diluição do mel, foi adicionado levedo de cerveja ao mosto, na concentração de 1 g L-1, como fonte adicional de nutrientes para as leveduras fermentativas. Para cada tipo de mel foram realizadas duas receitas, sendo uma com a fermentação ocorrendo de forma espontânea com os microrganismos naturalmente presente nos méis, e outra com a esterilização dos méis e adição de leveduras comerciais (Tabela 1). Para isso, o mosto preparado foi separado em duas alíquotas, sendo metade pasteurizado por aquecimento a 75 °C por 20 min. Após o resfriamento, o mosto pasteurizado foi utilizado para a composição das receitas submetidas à fermentação com leveduras comerciais. As receitas foram acondicionadas em câmaras para testes de demanda bioquímica de oxigênio (BOD) por 15 dias com uma temperatura constante de 19 °C. Após o processo de fermentação, as amostras foram envasadas em garrafas de vidro.
Análise da composição micromolecular de bebidas fermentadas e méis por Molecular Networking baseado em dados de CLAE-IES-EM/EM As amostras dos méis e de suas respectivas bebidas fermentadas foram analisadas por CLAE-IES-EM/EM, de modo a obter espectros EM/EM característicos de cada amostra para posterior criação de redes moleculares (Molecular Networking) que possibilitam comparação e anotação de compostos. Análises por CLAE-IES-EM/EM O preparo das amostras baseou-se no método descrito por Silva Júnior et al.,14 com modificações. Alíquotas de 260 mg das amostras dos méis foram transferidas para microtubos de 2,0 mL. Adicionou-se 1,8 mL de água em cada tubo, os quais foram submetidos à agitação em vórtex até a completa solubilização. Posteriormente, realizou-se a extração em fase sólida das amostras em cartuchos com a fase estacionária C18. Inicialmente, os cartuchos foram eluídos com 3 × 1 mL de água para o condicionamento. Depois, realizou-se a eluição da amostra e, em seguida, eluição com 3 × 1 mL de água. Por fim, realizou-se a eluição com 1,2 mL de metanol grau HPLC. A fração metanólica de cada amostra foi submetida ao concentrador à vácuo a 40 °C, por 4 h. Após a concentração, adicionou-se 150 µL de solução de ácido fórmico 0,1%:metanol grau HPLC (2:1) em cada amostra da fração metanólica e estas foram submetidas a agitação em vórtex até a completa solubilização. Alíquotas de 520 mg das bebidas fermentadas foram transferidas para microtubos de 2,0 mL. Adicionou-se 1,4 mL de água em cada tubo, os quais foram submetidos à agitação em vórtex até a completa solubilização. Em seguida, realizou-se extração em fase sólida das amostras das bebidas em cartuchos com a fase estacionária C18, seguindo procedimentos idênticos aos aplicados aos méis. As frações metanólicas das bebidas foram ressuspendidas em 150 µL de solução de ácido fórmico 0,1%:metanol grau HPLC (2:1) e filtradas com filtro de seringa de politetrafluoretileno (PTFE) (0,22 µm) para inserts em vials de 1,0 mL e analisadas por CLAE-IES-EM/EM. As análises foram realizadas em um cromatógrafo UFLC LC-6AD (Shimadzu), equipado com detector de arranjo de diodos (DAD) e acoplado ao espectrômetro de massas micrOTOF-QII (Bruker Daltonics). O método de CLAE foi desenvolvido em coluna C18 (LunaTM, Phenomenex), com dimensões de 250 × 4,6 mm e tamanho de partícula de 5 µm, conectada a uma pré-coluna com a mesma fase estacionária. A fase móvel foi composta por solução de ácido fórmico 0,1% (A) e acetonitrila (ACN) acrescida de 0,1% de ácido fórmico (B) ao fluxo de 1,0 mL min-1. Utilizou-se o seguinte gradiente de eluição: 0-5 min (3% B); 5-35 min (3-25% B); 35-50 min (3-70% B); 50-52 min (70-90% B); 52-56 min (90% B); 56-59 min (90-3%); 59-70 min (3% B). A temperatura da coluna foi de 40 °C. O volume de injeção foi de 20 µL. Os espectros de EM e EM2 foram adquiridos entre m/z 50 e 1500, usando-se ionização no modo positivo. Os parâmetros de IES-EM foram: voltagem do capilar de 3,5 kV; end plate offset de 450 V; temperatura de secagem de 350 °C; fluxo de gás de 10,0 L min-1 e pressão de 5,0 bar. Utilizou-se nitrogênio como gás de nebulização, de secagem e de colisão. Os espectros de EM/EM foram adquiridos no modo Auto MS/MS, realizando-se a fragmentação automática dos três íons mais intensos de cada scan (EM1). Foi realizada a infusão automática da solução de trifluoroacetato de sódio 400 mg L-1 ao final de cada análise para a calibração dos espectros em alta resolução. Para as fragmentações dos íons precursores, utilizou-se uma rampa de energia de colisão de 20 a 50 eV. Os dados obtidos foram analisados no software DataAnalysis 4.3 (Bruker Daltonik, Alemanha). Construção de rede molecular baseada em dados de CLAE-IES-EM/EM (molecular networking) O processamento em redes moleculares tem como objetivo o agrupamento de espectros EM/EM que apresentam fragmentações homólogas.19,20 Para construção de uma rede molecular, os dados de EM/EM são inicialmente alinhados como espectros de consenso de cada sinal. Estes espectros são projetados como nodos, os quais são conectados por arestas de acordo com suas similaridades de cosseno, em valor que varia de 0 a 1, sendo que 1 indica a maior similaridade ou identidade, e 0 indica totalmente diferentes.21 Consequentemente, estas redes permitem a visualização de conjuntos de espectros de estruturas relacionadas, mesmo nos casos em que estes espectros não correspondem a compostos conhecidos. Além disso, os espectros experimentais podem ser comparados aos de bibliotecas espectrais públicas da plataforma, o que permite anotações moleculares.22 Neste trabalho, utilizamos o GNPS (Global Natural Products Social Molecular Networking), uma plataforma online e de livre acesso que disponibiliza ferramentas para construção de redes moleculares (do inglês, molecular networking) a partir de dados de EM/EM.22,24 Para a construção das redes, os arquivos das análises por HPLC-MS/MS de todas as amostras de méis e produtos fermentados foram separadamente convertidos para o formato .mzML no software MSConvert, versão 3 (ProteoWizard Software Foundation, EUA).25 Em seguida, os arquivos .mzML foram submetidos ao pré-processamento com o software MZmine, versão 3.9.0 (BMC Bioinformatics, Reino Unido). Foram ajustados parâmetros relacionados à eliminação de ruído em espectros de EM1 e EM2, detecção de sinais cromatográficos, desconvolução (pareamento EM1-EM2), agrupamento de íons isótopos e alinhamento dos sinais entre os cromatogramas, utilizando os seguintes parâmetros e valores: (I) mass detection, mass detector centroid, MS level filter 1, noise level 1.0E3; (II) mass detection, mass detector centroid, MS level filter 2, noise level 2.0E1; (III) ADAP chromatogram builder, minimum consecutive scans 3, minimum intensity for consecutive scans 1.0E3; minimum absolute height 1.5E3, m/z tolerance (scan-to-scan) 0.002 m/z or 5 ppm; (IV) ADAP feature resolver, dimension - retention time, S/N threshold 7, S/N estimator - intensity SN, min feature height 1,000, coefficient/area threshold 7, peak duration range 0.02-2.00, RT wavelet range 0.02-0.02; (V) 13C isotope filter (formerly: isotope grouper), m/z tolerance (intra-sample) 0.002 m/z or 5 ppm, retention time tolerance 0.2 min, maximum charge: 2, representative isotope - most intense; (VI) join aligner, m/z tolerance (sample-to-sample) 0.02 m/z or 0 ppm, weight for m/z 50, retention time tolerance 0.3 min, weight for RT 50; (VI) filter list rows filter: marcação de never remove feature with MS2 e reset the feature number with ID. Ao final do pré-processamento, foram gerados um arquivo .mgf com as informações dos espectros de EM2, e um arquivo .csv com a matriz de dados que inclui os íons precursores, seus respectivos tempos de retenção (tR) e intensidades nos cromatogramas de cada amostra. Os arquivos .mgf e .csv foram carregados para plataforma GNPS226 e analisados pela ferramenta Feature-based Molecular Networking (FBMN).27 Um arquivo .txt, contendo uma tabela com os metadados (espécies e condições de fermentação) das amostras, também foi carregado para o GNPS2. Entre os parâmetros para construção da rede molecular, as tolerâncias de variação de m/z tanto para o íon precursor, quanto para o íon produto foram ajustadas para 0,02 Da. A conexão entre espectros de consenso foi ajustada para um valor mínimo de cosseno de 0,65 e o mínimo de 5 íons em comum. Ajustou-se o mínimo valor de cosseno de 0,7 e o mínimo de 5 íons em comum para correspondência entre um espectro experimental e o da biblioteca. Avaliação da composição dos voláteis presentes nos méis e bebidas fermentadas Análises por CG-EM As amostras de méis e fermentados foram submetidas à microextração em fase sólida (SPME, do inglês, solid phase micro extraction) no modo headspace. Neste procedimento, 300 mg das amostras de méis e 100 µL dos fermentados foram transferidos para frascos de 4 mL, os quais foram mantidos em banho-maria a 80 °C por 30 min. Durante este período, uma fibra para holder manual (50/30 μm divinilbenzeno/carboxeno/polidimetilsiloxano (DVB/CAR/PDMS), Supelco) de 2 cm foi exposta ao interior de cada frasco. Após a exposição e adsorção dos constituintes voláteis, a fibra foi aquecida na fonte do equipamento e analisada em um sistema CG-EM com ionização por elétrons a 70 eV, sendo utilizado um cromatógrafo modelo GCMS-QP2010 (Shimadzu) e coluna ZB-624 (Phenomenex) com dimensões de 30 m × 0,32 mm × 1,8 µm. As amostras foram analisadas em modo split (1:5) com tempo de dessorção da fibra de 5 min. O injetor e a fonte foram mantidos a 250 °C. Utilizou-se hélio como gás carreador a 2,04 mL min-1 e o seguinte gradiente de temperatura: 35 °C por 5 min, 35-240 °C a 6 °C min-1, e 240 °C por 15 min. Os constituintes foram anotados a partir de suas similaridades espectrais com os espectros disponíveis nas bibliotecas Wiley, National Institute of Standards and Technology (NIST) e Flavors and Fragrances of Natural and Synthetic Compounds (FFNSC). As informações podem ser consultadas no Material Suplementar.
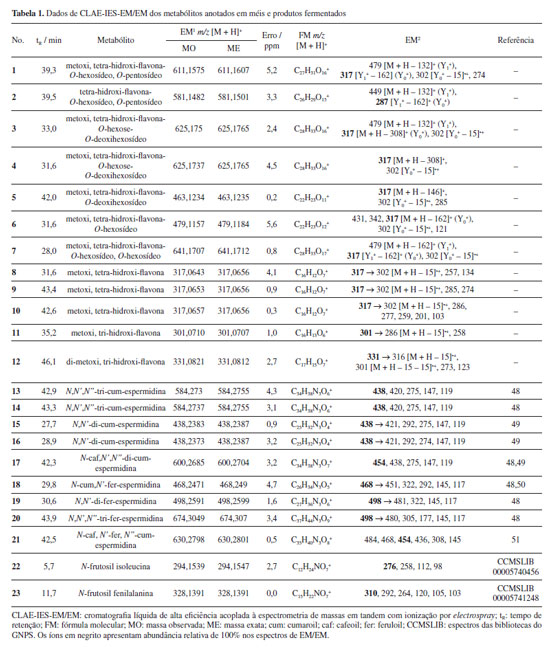
RESULTADOS E DISCUSSÃO Análise da composição micromolecular de bebidas fermentadas e méis por rede molecular (Molecular Networking) baseada em dados de CLAE-IES-EM/EM Os dados de CLAE-IES-EM/EM de méis e bebidas fermentadas foram processados no GNPS2 com a ferramenta FBMN, obtendo-se uma rede molecular. Informações para o acesso aos dados da rede molecular e aos dados brutos das análises por CLAE-IES-EM/EM encontram-se na seção "Declaração de Disponibilidade de Dados". Esta rede exibiu agrupamentos (clusters) de espectros de consenso anotados como flavonoides pelas bibliotecas do GNPS. A confirmação das anotações e propagação destas foi realizada a partir das sugestões estruturais das bibliotecas espectrais, inspeção manual dos dados de EM/EM e diferenças de massa entre nodos. Foram considerados como anotados os metabólitos que apresentaram íons correspondentes às massas exatas de suas respectivas fórmulas moleculares protonadas com erros inferiores a 10 ppm (Tabela 1). Nos perfis de fragmentação dos flavonoide-O-glicosídeos (1 a 7), foram verificadas perdas neutras relacionadas aos resíduos de açúcares, como pentose (-132 u), hexose (-162 u), hexose-deoxihexose (-308 u) e deoxihexose (-146 u). As reações de fragmentação que envolvem perdas de açúcares formam os íons Y0+, correspondentes a estrutura da aglicona livre, enquanto íons Y1+ podem ser observados em espectros de flavonoides ligados a dois açúcares (Tabela 1). Os íons m/z [Y0+ - 15]•+ referem-se a perdas de radical metil (•CH3), o que indica a presença de grupo metoxi em anel aromático28 e, dessa forma, flavonoides metoxilados, como os O-glicosídeos 1, 3 a 7, e as agliconas 8 a 12 (Tabela 1). No entanto, os dados espectrométricos não permitiram determinar as posições dos grupos metoxi, hidroxi e açúcares nos flavonoides. Os derivados hidroxicinâmicos de espermidina 13 a 21, também conhecidos como fenolamidas, foram anotados a partir da comparação de seus perfis de fragmentação com dados de EM2 disponíveis na literatura (Tabela 1). As fenolamidas são encontradas em plantas, principalmente em flores e pólen,29-32 sendo também detectadas nos ninhos de ASF.11 Observou-se também um cluster de derivados de aminoácidos, sendo que os aminoácidos ligados à açúcares 22 e 23 foram anotados a partir de matches espectrais com bibliotecas do GNPS2 (Tabela 1, Figura 1).
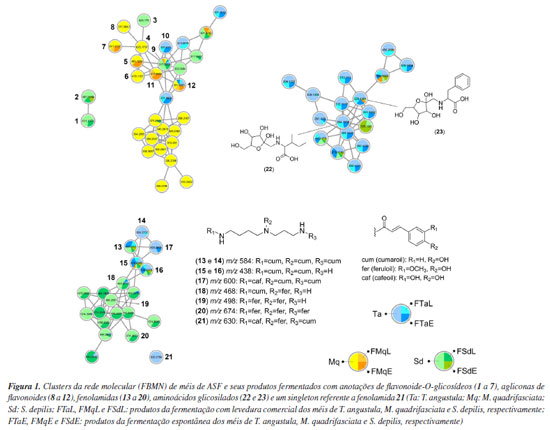
Verificou-se que, de forma geral, os flavonoides anotados foram encontrados tanto nos méis, quanto nas bebidas fermentadas (Figura 1), sugerindo que o processo de fermentação dos méis não apresenta influência sobre estes metabólitos. Os O-glicosídeos 1 e 2 foram detectados exclusivamente nas amostras de S. depilis, enquanto 5, 7 e 11 foram anotados somente em méis e fermentados de M. quadrifasciata, e a aglicona 10 foi encontrada somente nas amostras de T. angustula. Flavonoides C e O-glicosídeos, e na forma de agliconas metoxiladas foram anotados no mel de Tetragonisca fiebrigi coletado no sudeste da Argentina.33 No entanto, os flavonoides descritos em T. fiebrigi divergem dos anotados em T. angustula neste trabalho. Em adição, observou-se a presença de feniletilamidas no mel de T. fiebrigi, as quais também não foram detectadas na amostra de T. angustula. Estes dados sugerem influências ambientais sobre a quimiodiversidade encontrada em méis de um mesmo gênero de abelhas, demonstrando que amostras de méis de regiões específicas podem apresentar assinaturas químicas únicas. Um trabalho prévio34 do nosso grupo associou as técnicas de CLAE-IES-EM/EM e CG-EM, e molecular networking para caracterização química da geoprópolis de M. quadrisfasciata, notando-se a presença do ácido dehidroabiético e diterpenos derivados entre os principais constituintes. Diferentemente, no mel de M. quadrisfasciata analisado no presente estudo, verificou-se a presença de flavonoides na forma glicosilada e de agliconas. Assim, os resultados obtidos evidenciam a diversidade de metabólitos associados aos diferentes produtos de uma mesma espécie de ASF. As fenolamidas (13 a 20) também foram detectadas em méis e bebidas fermentadas, principalmente nas amostras das espécies T. angustula e S. depilis. Notou-se ainda que a ocorrência das fenolamidas 18, 19 e 20 foi mais restrita ao mel de S. depilis e seu produto de fermentação com levedura comercial (Figura 1). Adicionalmente, estes dados contribuem para o conhecimento sobre a composição micromolecular de méis de abelhas nativas do Brasil, bem como diferenças na ocorrência de metabólitos em méis de diferentes espécies de ASF. Compostos fenólicos encontrados em plantas, como derivados do ácido cinâmico e flavonoides, possuem alta capacidade de eliminação de radicais livres e são potenciais moléculas para prevenção e tratamento de enfermidades relacionadas ao estresse oxidativo, como doenças cardiovasculares, neurodegenerativas, câncer e diabetes.35 Os méis de ASF, por sua vez, possuem grande potencial antioxidante, até três vezes maior do que o mel de abelhas européias.36 Não somente o conteúdo total de compostos fenólicos reflete o potencial antioxidante do mel, mas também a sua composição específica.37 Por exemplo, a atividade antioxidante de fenolamida com três grupos cumaroil (13 e 14) foi observada in vitro.38 A grande diversidade de compostos fenólicos identificada pelas técnicas empregadas no presente trabalho abre uma frente importante de conhecimento, referente não só ao potencial quantitativo (concentração relativa) desses compostos, mas também aos aspectos qualitativos como composição e diversidade. Além da grande atividade antioxidante, os flavonoides apresentam propriedades anticâncer, anti-inflamatória, antibacteriana, antifúngica, antiviral, tripanocida, leishmanicida, moluscicida, bem como efeitos cardio e neuroprotetores, o que demonstra os benefícios de sua ingestão para saúde humana.39-44 Avanços biotecnológicos para a aplicação desses compostos em produtos nutracêuticos e farmacêuticos vêm sendo conquistados, principalmente visando otimizar a biodisponibilidade e produção em escala. O desenvolvimento dessas moléculas para uso em tratamentos de saúde é bastante atrativo, visto a gama de benefícios à saúde e aparente ausência de efeitos adversos.45 Entretanto, mais estudos, principalmente envolvendo testes clínicos, ainda se fazem necessários para a maioria dos compostos, além de testes de farmacocinética, determinação de dose e possíveis interações medicamentosas, para que as aplicações com benefícios para a saúde humana sejam amplamente alcançadas.46 Embora a abundância natural dos compostos fenólicos seja frequentemente baixa, estratégias biotecnológicas, incluindo a síntese enzimática, estão sendo exploradas para superar as limitações de produção e facilitar sua incorporação em formulações de alimentos funcionais e farmacêuticas.47 Avaliação da composição dos voláteis presentes nos méis e bebidas fermentadas Os perfis cromatográficos dos méis das três espécies obtidos por CG-EM mostraram a presença de voláteis de diferentes classes químicas (Figuras 2-4). Os constituintes majoritários dos méis de T. angustula (Ta) e M. quadrifasciata (Mq) são o acetato de etila (24), o ácido acético (26) e um ácido graxo (55) (Figuras 2 e 3). O mel de S. depilis (Sd) apresentou a maior variedade de constituintes entre os méis analisados, sendo que os sinais majoritários do cromatograma correspondem a ésteres (35, 57 e 59), ácidos (26, 40 e 55 ) e álcoois (42, 43, 61 e 63). Estes dados mostram que, além de variações na ocorrência de derivados fenólicos, amostras de méis de espécies distintas também apresentaram diferenças na presença de voláteis.
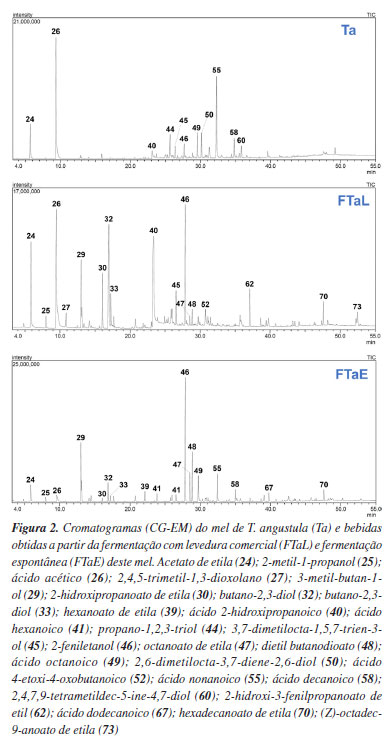
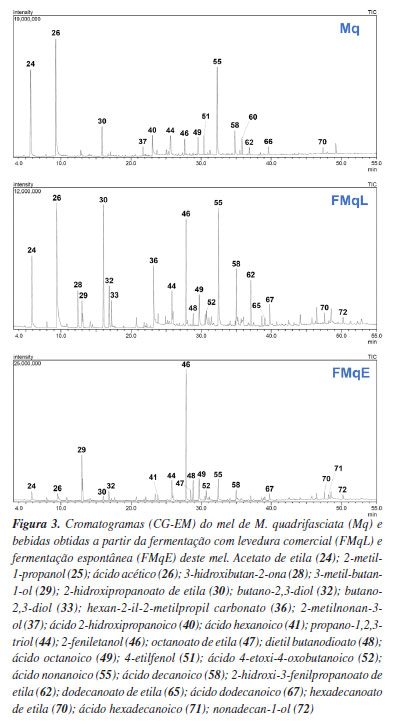
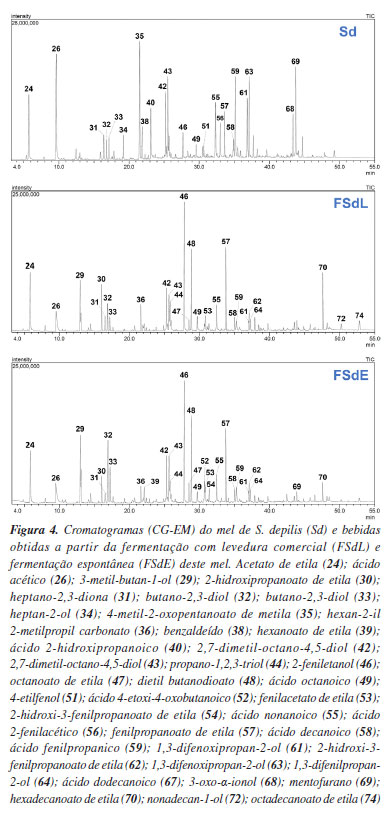
As Figuras 2-4 evidenciam de forma clara as diferenças entre os perfis cromatográficos dos méis in natura e de suas respectivas bebidas fermentadas. No caso do mel de T. angustula, os constituintes majoritários antes da fermentação (24, 26 e 55) são substituídos, após o processo fermentativo, pelo predomínio de álcoois (29, 32, 33, 46) e ésteres (30, 47, 48, 62, 70, 73), destacando-se o 2-feniletanol (46), associado ao aroma floral, e o ácido lático (40), responsável por notas sensoriais ácidas. De forma semelhante, no mel de M. quadrifasciata, os principais constituintes iniciais (24, 26 e 55) são sucedidos nos fermentados por álcoois de cadeia curta e média (29, 32, 33, 44, 46) e diferentes ésteres (30, 48, 62, 70), com destaque para o 2-feniletanol (46) e o 2-hidroxipropanoato de etila, que conferem notas florais e frutadas, respectivamente. Já no mel de S. depilis, observa-se uma maior diversidade inicial de constituintes (35, 57, 59, 26, 40, 55, 42, 43, 61, 63), que após a fermentação sofrem alterações marcantes, com intensificação de álcoois (29, 32, 33, 44, 46) e ésteres (30, 47, 48, 57, 62, 70), evidenciando a forte influência da atividade fermentativa sobre a composição volátil. De modo geral, a intensificação de álcoois (29, 32, 33, 44, 46) e do ácido lático (40) adiciona à camada olfativa das bebidas fermentadas características típicas da fermentação, incluindo notas alcoólicas e ácidas. Além disso, a maior diversidade e intensidade de ésteres (30, 47, 48, 57, 62, 70, 73) contribui para o aumento da complexidade aromática, resultando em bebidas mais ricas em nuances sensoriais em comparação aos méis in natura. Além disso, as diferenças entre os perfis cromatográficos de produtos obtidos a partir de fermentações espontâneas (FTaE e FMqE) e com leveduras comerciais (FTaL e FMqL) evidenciam a influência do processo fermentativo sobre a composição química das bebidas de T. angustula e M. quadrifasciata (Figuras 3 e 4). Nos fermentados com levedura comercial observa-se maior intensidade de compostos como o ácido lático (40) e o 2-feniletanol (46), que conferem notas ácidas e florais mais pronunciadas, enquanto nas fermentações espontâneas aparecem ésteres adicionais (por exemplo, 57 e 62), associados a nuances frutadas mais complexas. Em contrapartida, as diferenças entre os cromatogramas dos fermentados de S. depilis (FSdL e FSdE) foram menos expressivas (Figura 4), sugerindo que, nessa espécie, a composição inicial mais diversa do mel exerce papel dominante sobre o perfil final da bebida. Esses resultados reforçam que tanto a composição do mel de origem quanto a comunidade microbiana responsável pela fermentação são determinantes na definição do perfil sensorial das bebidas resultantes. As receitas FTaL, FMqL, FSdL e FSdE apresentaram altas intensidades dos sinais correspondentes ao acetato de etila (24) e ácido acético (26) em seus cromatogramas, sendo consideradas inapropriadas para o consumo (Figuras 2-4). Assim, a análise dos compostos voláteis resultantes da fermentação das diferentes amostras de méis forneceu informações acerca da diversidade sensorial e segurança alimentar das bebidas obtidas. A diversidade sensorial dos méis, apontada pelas técnicas aqui utilizadas, abre caminhos para a aplicação industrial dos voláteis identificados como aromatizantes naturais, levando sabores mais complexos e inovadores para a indústria alimentícia.
CONCLUSÕES Os méis e hidroméis analisados apresentaram um perfil metabolômico complexo, com flavonoides e ampla diversidade de compostos voláteis. A presença de flavonoides, conhecidos por seu potencial antioxidante, sugere benefícios à saúde. Esses compostos foram detectados tanto nos méis quanto nas bebidas fermentadas, indicando que o processo de fabricação do hidromel preserva suas propriedades bioativas. Foram observadas especificidades quanto à composição de flavonoides em cada amostra de mel, com compostos exclusivos de cada espécie, como por exemplo a aglicona (10), encontrada somente nas amostras de T. angustula. O mel de S. depilis, por sua vez, foi o que apresentou maior diversidade de compostos. Tais características podem resultar em méis com diferentes propriedades bioativas de acordo com a espécie ou região geográfica. Com relação ao perfil sensorial dos méis e hidroméis, a análise de compostos voláteis apontou diferenças, principalmente pela presença de álcoois e ésteres nas bebidas fermentadas, como esperado. Ainda com relação ao processo de fermentação, foram apontadas diferenças quanto às bebidas produzidas com leveduras comerciais ou por fermentação espontânea, indicando composições químicas, e consequentemente, experiências sensoriais distintas para cada processo fermentativo. Os resultados mostram que os hidroméis de ASF são bebidas com aromas e sabores diversificados e complexos, como evidenciado pela análise de compostos voláteis, e com potencial efeito benéfico à saúde por serem ricos em flavonoides. Além de seu consumo em bebidas, os compostos identificados apresentam ampla aplicabilidade industrial, incluindo o desenvolvimento de alimentos funcionais e produtos farmacêuticos com propriedades antioxidantes, anti-inflamatórias e protetoras contra doenças crônicas. Ao mesmo tempo, os hidroméis trazem inovação ao mercado, tanto pelo seu caráter inédito quanto pela valorização da biodiversidade brasileira.
MATERIAL SUPLEMENTAR O material suplementar desse trabalho (dados dos constituintes voláteis anotados a partir das análises por CG-EM) está disponível em http://quimicanova.sbq.org.br/,na forma de arquivo PDF, com acesso livre.
DECLARAÇÃO DE DISPONIBILIDADE DE DADOS A rede molecular construída a partir do processamento dos dados de CLAE-IES-EM/EM no GNPS2 com a ferramenta FBMN está disponível em https://gnps2.org/status?task=c0de2e0e1f8146e48cc15f462e6ce502. Os dados brutos das análises por CLAE-IES-EM/EM estão disponíveis no repositório MassIVE (MSV000097990) e podem ser acessados em https://massive.ucsd.edu/ProteoSAFe/dataset.jsp?task=5e2f3a5b0aa64fbbb2131dad5f3016d6.
AGRADECIMENTOS Os autores agradecem à Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP, 2020/02207-5, 2025/02994-0 e 2019/22357-4) pelo apoio financeiro, projeto PIPE FAPESP (2022/04784-5), à Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES) e ao Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq).
REFERÊNCIAS 1. Nogueira-Neto, P.; Vida e Criação de Abelhas Indígenas sem Ferrão, 3ª ed.; Nogueirapis: São Paulo, 1997. 2. Menezes, C.; Alves, D. A.; Lucena, D. A. A.; Almeida, E. A. B.; Abelhas sem Ferrão Relevantes para Meliponicultura no Brasil; Abelha: São Paulo, 2023. 3. Hrncir, M.; Jarau, S.; Barth, F. G.; J. Comp. Physiol., A 2016, 202, 597. [Crossref] 4. Pedro, S. R. M.; Sociobiology 2014, 61, 348. [Crossref] 5. Crane, E.; Honey: A Comprehensive Survey, 1st ed.; William Heinemann: London, 1975. 6. Gheldof, N.; Wang, X. H.; Engeseth, N. J.; J. Agric. Food Chem. 2002, 50, 5870. [Crossref] 7. Israili, Z. H.; Am. J. Ther. 2014, 21, 304. [Crossref] 8. Kumar, S.; Pandey, A. K.; Sci. World J. 2013, 2013, 1. [Crossref] 9. Forbes Brasil, Abelhas Nativas se tornam Rainhas na Alta Gastronomia de Badalados Restaurantes, https://forbes.com.br/forbesagro/2021/05/abelhas-nativas-se-tornam-rainhas-na-alta-gastronomia-de-badalados-restaurantes, acessado em setembro 2025. 10. Souza, B. A.; Marchini, L. C.; Dias, C. T. S.; Oda-Souza, M.; Carvalho, C. A. L.; Alves, R. M. O.; Cienc. Tecnol. Aliment. (Campinas, Braz.) 2009, 29, 798. [Crossref] 11. Nordin, A.; Sainik, N. Q. A. V.; Chowdhury, S. R.; Saim, A. B.; Idrus, R. B. H.; J. Food Compos. Anal. 2018, 73, 91. [Crossref] 12. Ávila, S.; Beux, M. R.; Ribani, R. H.; Zambiazi, R. C.; Trends Food Sci. Technol. 2018, 81, 37. [Crossref] 13. Sousa, J. P. L. M.: Perfil Físico-Químico, Composição Química e Capacidade Antioxidante de Méis Produzidos por Abelhas sem Ferrão; Dissertação de Mestrado, Universidade Federal Rural do Rio de Janeiro, Seropédica, Brasil, 2014. [Link] acessado em setembro 2025 14. Silva Junior, E. A.; Paludo, C. R.; Amaral, J. G.; Gallon, M. E.; Gobbo-Neto, L.; Nascimento, F. S.; Lopes, N. P.; ACS Omega 2019, 4, 15208. [Crossref] 15. McGovern, P. E.; Zhang, J.; Tang, J.; Zhang, Z.; Hall, G. R.; Moreau, R. A.; Nuñez, A.; Butrym, E. D.; Richards, M. P.; Wang, C. S.; Cheng, G.; Zhao, Z.; Wang, C.; Proc. Natl. Acad. Sci. U. S. A. 2004, 101, 17593. [Crossref] 16. Schrammm, K.; The Compleat Meadmaker: Home Production of Honey Wine from your First Batch to Award-Winning Fruit and Herb Variations; Brewers Publications: Boulder, 2003. 17. Reitenbach, A. F.; Lorenzi, A. S.; Ghesti, G. F.; dos Santos, P. C. M.; Rodrigues, I. M. T.; Barbosa, A. C.; Sant'Ana, R. R. A.; Fritzen-Freire, C. B.; Nowruzi, B.; Burin, V. M.; Fermentation 2025, 11, 226. [Crossref] 18. American Mead Makers Association, About AMMA: Mead's Revival in the US, https://mead-makers.org/about/, acessado em setembro 2025. 19. Olivon, F.; Allard, P. M.; Koval, A.; Righi, D.; Genta-Jouve, G.; Neyts, J.; Apel, C.; Pannecouque, C.; Nothias, L. F.; Cachet, X.; Marcourt, L.; Roussi, F.; Katanaev, V. L.; Touboul, D.; Wolfender, J. L.; Litaudon, M.; ACS Chem. Biol. 2017, 12, 2644. [Crossref] 20. Yang, J. Y.; Sanchez, L. M.; Rath, C. M.; Liu, X.; Boudreau, P. D.; Bruns, N.; Glukhov, E.; Wodtke, A.; De Felicio, R.; Fenner, A.; Wong, W. R.; Linington, R. G.; Zhang, L.; Debonsi, H. M.; Gerwick, W. H.; Dorrestein, P. C.; J. Nat. Prod. 2013, 76, 1686. [Crossref] 21. Pilon, A. C.; Vieira, N. C.; Amaral, J. G.; Monteiro, A. F.; Ricardo, R.; Spíndola, L. S, Castro-Gamboa, I.; Lopes, N. P.; Quim. Nova 2021, 44, 1168. [Crossref] 22. Wang, M.; Carver, J. J.; Phelan, V. V.; Sanchez, L. M.; Garg, N.; Peng, Y.; Nguyen, D. D.; Watrous, J.; Kapono, C. A.; Luzzatto-Knaan, T.; Porto, C.; Bouslimani, A.; Melnik, A. V.; Meehan, M. J.; Liu, W. T.; Crüsemann, M.; Boudreau, P. D.; Esquenazi, E.; Sandoval-Calderón, M.; Kersten, R. D.; Pace, L. A.; Quinn, R. A.; Duncan, K. R.; Hsu, C. C.; Floros, D. J.; Gavilan, R. G.; Kleigrewe, K.; Northen, T.; Dutton, R. J.; Parrot, D.; Carlson, E. E.; Aigle, B.; Michelsen, C. F.; Jelsbak, L.; Sohlenkamp, C.; Pevzner, P.; Edlund, A.; McLean, J.; Piel, J.; Murphy, B. T.; Gerwick, L.; Liaw, C. C.; Yang, Y. L.; Humpf, H. U.; Maansson, M.; Keyzers, R. A.; Sims, A. C.; Johnson, A. R.; Sidebottom, A. M.; Sedio, B. E.; Klitgaard, A.; Larson, C. B.; Boya, C. A. P.; Torres-Mendoza, D.; Gonzalez, D. J.; Silva, D. B.; Marques, L. M.; Demarque, D. P.; Pociute, E.; O'Neill, E. C.; Briand, E.; Helfrich, E. J. N.; Granatosky, E. A.; Glukhov, E.; Ryffel, F.; Houson, H.; Mohimani, H.; Kharbush, J. J.; Zeng, Y.; Vorholt, J. A.; Kurita, K. L.; Charusanti, P.; McPhail, K. L.; Nielsen, K. F.; Vuong, L.; Elfeki, M.; Traxler, M. F.; Engene, N.; Koyama, N.; Vining, O. B.; Baric, R.; Silva, R. R.; Mascuch, S. J.; Tomasi, S.; Jenkins, S.; Macherla, V.; Hoffman, T.; Agarwal, V.; Williams, P. G.; Dai, J.; Neupane, R.; Gurr, J.; Rodríguez, A. M. C.; Lamsa, A.; Zhang, C.; Dorrestein, K.; Duggan, B. M.; Almaliti, J.; Allard, P. M.; Phapale, P.; Nothias, L. F.; Alexandrov, T.; Litaudon, M.; Wolfender, J. L.; Kyle, J. E.; Metz, T. O.; Peryea, T.; Nguyen, D. T.; VanLeer, D.; Shinn, P.; Jadhav, A.; Müller, R.; Waters, K. M.; Shi, W.; Liu, X.; Zhang, L.; Knight, R.; Jensen, P. R.; Palsson, B.; Pogliano, K.; Linington, R. G.; Gutiérrez, M.; Lopes, N. P.; Gerwick, W. H.; Moore, B. S.; Dorrestein, P. C.; Bandeira, N.; Nat. Biotechnol. 2016, 34, 828 [Crossref]; Global Natural Products Social (GNPS), https://gnps.ucsd.edu, acessado em setembro 2025. 23. Zuffa, S.; Schmid, R.; Bauermeister, A.; Gomes, P. W. P.; Caraballo‑Rodriguez, A. M.; El Abiead, Y.; Aron, A. T.; Gentry, E. C.; Zemlin, J.; Meehan, M. J.; Avalon, N. E.; Cichewicz, R. H.; Buzun, E.; Terrazas, M. C.; Hsu, C. Y.; Oles, R.; Ayala, A. V.; Zhao, J.; Chu, H.; Kuijpers, M. C. M.; Jackrel, S. L.; Tugizimana, F.; Nephali, L. P.; Dubery, I. A.; Madala, N. E.; Moreira, E. A.; Costa-Lotufo, L. V.; Lopes, N. P.; Teixeira, P. R.; Jimenez, P. C.; Rimal, B.; Patterson, A. D.; Traxler, M. F.; Pessotti, R. C.; Villalobos, D. A.; Tamayo-Castillo, G.; Chaverri, P.; Escudero-Leyva, E.; Quiros-Guerrero, L. M.; Bory, A. J.; Joubert, J.; Rutz, A.; Wolfender, J. L.; Allard, P. M.; Sichert, A.; Pontrelli, S.; Pullman, B. S.; Bandeira, N.; Gerwick, W. H.; Gindro, K.; Massana-Codina, J.; Wagner, B. C.; Forchhammer, K.; Petras, D.; Aiosa, N.; Garg, N.; Liebeke, M.; Bourceau, P.; Kang, K. B.; Gadhavi, H.; de Carvalho, L. P. S.; dos Santos, M. S.; Pérez-Lorente, A. I.; Molina‑Santiago, C.; Romero, D.; Franke, R.; Brönstrup, M.; de León, A. V. P.; Pope, P. B.; La Rosa, S. L.; La Barbera, G.; Roager, H. M.; Laursen, M. F.; Hammerle, F.; Siewert, B.; Nat. Microbiol. 2024, 9, 336. [Crossref] 24. Gauglitz, J. M.; West, K. A.; Bittremieux, W.; Williams, C. L.; Weldon, K. C.; Panitchpakdi, M.; Di Ottavio, F.; Aceves, C. M.; Brown, E.; Sikora, N. C.; Jarmusch, A. K.; Cameron Martino, Tripathi, A.; Meehan, M. J.; Dorrestein, K.; Shaffer, J. P.; Coras, R.; Vargas, F.; Goldasich, L. D.; Schwartz, T.; Bryant, M.; Humphrey, G.; Johnson, A. J.; Spengler, K.; Dorrestein, P. C.; Nat. Biotechnol. 2022, 40, 1774. [Crossref] 25. Chambers, M. C.; Maclean, B.; Burke, R.; Amodei, D.; Ruderman, D. L.; Neumann, S.; Gatto, L.; Fischer, B.; Pratt, B.; Egertson, J.; Hoff, K.; Kessner, D.; Tasman, N.; Shulman, N.; Frewen, B.; Baker, T. A.; Brusniak, M. Y.; Paulse, C.; Creasy, D.; Flashner, L.; Kani, K.; Moulding, C.; Seymour, S. L.; Nuwaysir, L. M.; Lefebvre, B.; Kuhlmann, F.; Roark, J.; Rainer, P.; Detlev, S.; Hemenway, T.; Huhmer, A.; Langridge, J.; Connolly, B.; Chadick, T.; Holly, K.; Eckels, J.; Deutsch, E. W.; Moritz, R. L.; Katz, J. E.; Agus, D. B.; MacCoss, M.; Tabb, D. L.; Nat. Biotechnol. 2012, 30, 918. [Crossref] 26. GNPS2, https://gnps2.org/homepage, acessado em setembro 2025. 27. Nothias, L. F.; Petras, D.; Schmid, R.; Dührkop, K.; Rainer, J.; Sarvepalli, A.; Protsyuk, I.; Ernst, M.; Tsugawa, H.; Fleischauer, M.; Aicheler, F.; Aksenov, A. A.; Alka, O.; Allard, P. M.; Barsch, A.; Cachet, X.; Caraballo-Rodriguez, A. M.; da Silva, R. R.; Dang, T.; Garg, N.; Gauglitz, J. M.; Gurevich, A.; Isaac, G.; Jarmusch, A. K.; Kameník, Z.; Kang, K. Bin; Kessler, N.; Koester, I.; Korf, A.; Le Gouellec, A.; Ludwig, M.; Martin H, C.; McCall, L. I.; McSayles, J.; Meyer, S. W.; Mohimani, H.; Morsy, M.; Moyne, O.; Neumann, S.; Neuweger, H.; Nguyen, N. H.; Nothias-Esposito, M.; Paolini, J.; Phelan, V. V.; Pluskal, T.; Quinn, R. A.; Rogers, S.; Shrestha, B.; Tripathi, A.; van der Hooft, J. J. J.; Vargas, F.; Weldon, K. C.; Witting, M.; Yang, H.; Zhang, Z.; Zubeil, F.; Kohlbacher, O.; Böcker, S.; Alexandrov, T.; Bandeira, N.; Wang, M.; Dorrestein, P. C.; Nat. Methods 2020, 17, 905. [Crossref] 28. Gates, P. J.; Lopes, N. P.; Int. J. Anal. Chem. 2012, 2012, 1. [Crossref] 29. Roumani, M.; Besseau, S.; Gagneul, D.; Robin, C.; Larbat, R.; J. Exp. Bot. 2021, 72, 2334. [Crossref] 30. Caldas, F. R. L.; Augusto Filho, F.; Facundo, H. T.; Alves, R. F.; dos Santos, F. D. A. R.; da Silva, G. R.; Camara, C. A.; Silva, T. M. S.; Quim. Nova 2019, 42, 49. [Crossref] 31. Negri, G.; Teixeira, E. W.; Alves, M. L. T. M. F.; Moreti, A. C. C. C.; Otsuk, I. P.; Borguini, R. G.; Salatino, A.; J. Agric. Food Chem. 2011, 59, 5516. [Crossref] 32. Sobolev, V. S.; Sy, A. A.; Gloer, J. B.; J. Agric. Food Chem. 2008, 56, 2960. [Crossref] 33. Dallagnol, A. M.; Dallagnol, V. C.; Vignolo, G. M.; Lopes, N. P.; Brunetti, A. E.; J. Agric. Food Chem. 2022, 70, 12596. [Crossref] 34. Feitosa, L. G. P.; Feres, J. M.; Godinho, C. C.; Albernaz, L. C.; Espindola, L. S.; Vessecchi, R.; Guaratini, T.; Lopes, N. P.; Rapid Commun. Mass Spectrom. 2025, 39, e10025. [Crossref] 35. Li, A. N.; Li, S.; Zhang, Y. J.; Xu, X. R.; Chen, Y. M.; Li, H.; Nutrients 2014, 6, 6020. [Crossref] 36. Shamsudin, S.; Selamat, J.; Sanny, M.; Bahari, A. R. S.; Jambari, N. N.; Khatib, A.; Molecules 2019, 24, 3898. [Crossref] 37. Majid, M.; Ellulu, M. S.; Abu Bakar, M. F.; Scientifica 2020, 2020, ID2529592. [Crossref] 38. Otha, S.; Fujimaki, T.; Uy, M. M.; Yanai, M.; Yukiyoshi, A.; Nat. Prod. Res. 2007, 21, 726. [Crossref] 39. Rodríguez-García, C.; Sánchez-Quesada, C.; Gaforio, J. J.; Gaforio, J. J.; Antioxidants 2019, 8, 137. [Crossref] 40. Ullah, A.; Munir, S.; Badshah, S. L.; Khan, N.; Ghani, L.; Poulson, B. G.; Emwas, A. H.; Jaremko, M.; Molecules 2020, 25, 5243. [Crossref] 41. Dias, M. C.; Pinto, D. C. G. A.; Silva, A. M. S.; Molecules 2021, 26, 5377. [Crossref] 42. Al-Khayri, J. M.; Sahana, G. R.; Nagella, P.; Joseph, B. V.; Alessa, F. M.; Al-Mssallem, M. Q.; Molecules 2022, 27, 2901. [Crossref] 43. Ross, J. A.; Kasum, C. M.; Annu. Rev. Nutr. 2002, 22, 19. [Crossref] 44. Keles, L. C.; Melo, N. I.; Aguiar, G. P.; Wakabayashi, A. L.; Carvalho, C. E.; Cunha W. R.; Crotti, A. E. M.; Quim. Nova 2010, 33, 2245. [Crossref] 45. Gervasi, T.; Calderaro, A.; Barreca, D.; Tellone, E.; Trombetta, D.; Ficarra, S.; Smeriglio, A.; Mandalari, G.; Gattuso, G.; Int. J. Mol. Sci. 2022, 23, 1710. [Crossref] 46. Hasnat, H.; Shompa, S. A.; Islam, M. M.; Alam, S.; Richi, F. T.; Emon, N. U.; Ashrafi, S.; Ahmed, N. U.; Chowdhury, M. N. R.; Fatema, N.; Hossain, M. S.; Ghosh, A.; Ahmed, F.; Heliyon 2024, 10, e27533. [Crossref] 47. Zeng, F.; Zhang, H.; Xu, M.; Huang, K.; Zhang, T.; Duan, J.; J. Biotechnol. 2021, 334, 51. [Crossref] 48. Handrick, V.; Vogt, T.; Frolov, A.; Anal. Bioanal. Chem. 2010, 398, 2789. [Crossref] 49. Xie, G.; Xu, Q.; Li, R.; Shi, L.; Han, Y.; Zhu, Y.; Wu, G.; Qin, M.; J. Pharm. Biomed. Anal. 2019, 164, 283. [Crossref] 50. Silva, A. B.; Duarte, N.; Mecha, E.; Belo, M.; Patto, M. C. V.; Bronze, M. R.; Foods 2020, 9, 14. [Crossref] 51. Negri, G.; Barreto, L. M. R. C.; Sper, F. L.; Carvalho, C.; Campos, M. G. R.; Brazilian J. Food Technol. 2018, 21, e2016176. [Crossref] |
On-line version ISSN 1678-7064 Printed version ISSN 0100-4042
Qu�mica Nova
Publica��es da Sociedade Brasileira de Qu�mica
Caixa Postal: 26037
05513-970 S�o Paulo - SP
Tel/Fax: +55.11.3032.2299/+55.11.3814.3602
Free access